U-Net 3+: 全尺度的跳跃连接的 UNet
点击上方“3D视觉工坊”,选择“星标”
干货第一时间送达

前言:U-Net由Olaf Ronneberger等人在在2015年MICCAI上提出。U-Net在神经元结构分割方面取得了巨大的成功,由于功能在层之间传播,因此其框架是突破性的。后续在U-Net的基础上涌现了许多优秀的架构如:U-Net++,Attention U-Net,U2-Net等,今天我们将介绍新的U-Net结构:U-Net+++。
通过增强U-Net架构,在多个数据集上U-NET 3+性能优于Attention UNET,PSPNet,DeepLabV2,DeepLabV3和DeepLabv3 +。这是发表2020 ICASSP的一篇论文,UNet++使用嵌套和密集跳过连接,但它没有从全尺度探索足够的信息。在 UNet 3+ 中,使用了全面的跳过连接和深度监督:
全尺度跳跃连接:将来自不同尺度特征图的低级细节与高级语义结合起来。
全尺度的深度监督:从全尺度聚合特征图中学习分层表示。
进一步提出了混合损失函数和分类引导模块(CGM)
UNet 3+提供更少的参数,但可以产生更准确的位置感知和边界增强的分割图。
论文:UNet 3+: A Full-Scale Connected UNet for Medical Image Segmentation
论文链接:https://arxiv.org/abs/2004.08790
代码链接:https://github.com/ZJUGiveLab/UNet-Version
01
全尺度跳跃连接
U-Net,U-Net++, U-Net3+ 结构图:
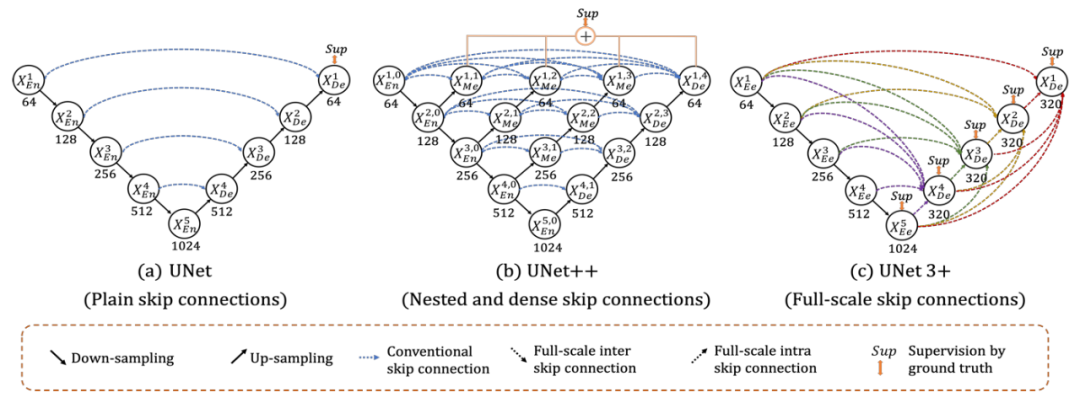
左:UNet,中UNet++,右:UNet 3+
无论是普通的连接U-Net还是密集连接的U-Net ++都缺乏从全尺度探索足够信息的能力,因此不能明确地得知器官的位置和边界。
U-Net 3+ 中的每个解码器层都融合了来自编码器的较小和相同尺度的特征图以及来自解码器的较大尺度的特征图,它们捕获了全尺度下的细粒度语义和粗粒度语义。
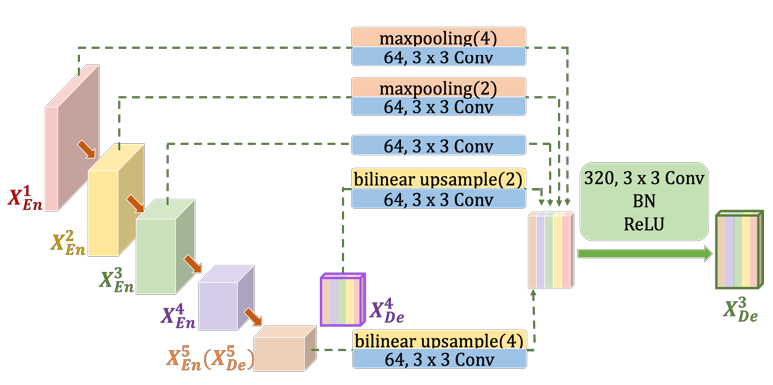
全尺度跳跃连接示例
为了构建 特征图,类似于U-NET,直接接收来自相同尺度编码器层的特征图 。
与U-Net 相比,一组编码器-解码间跳跃连接通过非重叠最大池化操作将来自较小尺度编码器层 和 进行池化下采样,以便传递底层的低级语义信息。
通过一系列内部解码器跳跃连接利用双线性插值传输来自大尺度解码器层 和 的高级语义信息。
UNet 3+ 中的参数少于UNet和UNet++ 中的参数。(这里有数学证明,有兴趣的可以阅读论文。)
02
全尺度的深度监督
2.1 深度监督
UNet 3+ 从每个解码器阶段(Sup1 到 Sup5)产生一个切分侧边输出,由真实的分类结果监督。
为了实现深度监督,每个解码器阶段的最后一层被送入一个普通的 3 × 3 卷积层,然后是一个双线性上采样和一个 sigmoid 函数。
2.2 损失函数
多尺度结构相似性指数 (MM-SSIM) 损失用于为模糊边界分配更高的权重。
Focal loss起源于RetinaNet,用于处理类不平衡问题。
使用标准IoU 损失。
因此,开发了一种混合损失用于在三级层次结构(像素级、补丁级和图片级)中进行分割,它能捕获大尺度的和精细结构清晰的边界,混合分割损失被定义为:
2.3. 分类引导模块 (CGM)
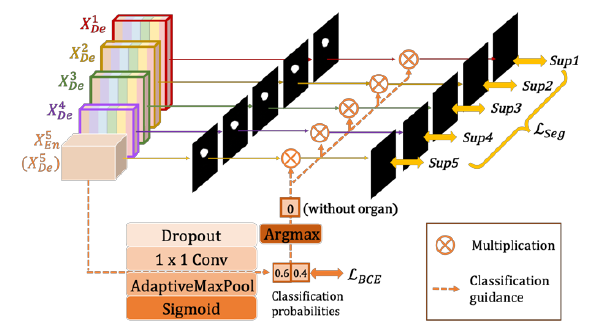
具有分类引导模块 (CGM) 的全面深度监督
非器官图像中存在误报。这可能是由于来自背景的噪声信息保留在较浅层中,导致过度分割现象。
为了解决这个问题,增加了一个额外的分类任务,用于预测输入图像是否有器官。如在上面的图所示,经过一系列的操作包括dropout,卷积,maxpooling,sigmoid, 最后有两个值代表有/没有器官的概率,最深层次的二维张量 ,其中每一个代表的有/无器官的概率。
在argmax 函数的帮助下,二维张量被转换为 {0,1} 的单个输出,表示有/无器官。
随后,单分类输出与侧分割输出相乘。由于二值分类任务的简单性,该模块通过优化二值交叉熵损失函数,轻松获得准确的分类结果,实现了对非器官图像过分割的指导。
03
实验结果
3.1 Datasets
肝脏分割数据集来自ISBI LiTS 2017 Challenge。它包含 131 幅对比增强的 3D 腹部 CT 扫描,其中 103 卷和 28 卷分别用于训练和测试。
来自医院的脾脏数据集,包含 40 和 9 个 CT 卷用于训练和测试。
图像被裁剪为 320×320。
3.2 与UNet和UNet++的比较
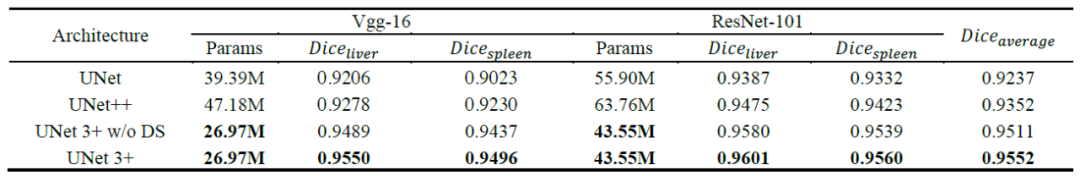
肝脏和脾脏数据集
测试了VGGNet和ResNet主干。没有深度监督的 UNet 3+ 实现了超越UNet和UNet++ 的性能,在两个数据集上执行的两个主干之间获得了 2.7 和 1.6 点的平均改进。UNet 3+结合全面深度监督进一步提升0.4分。
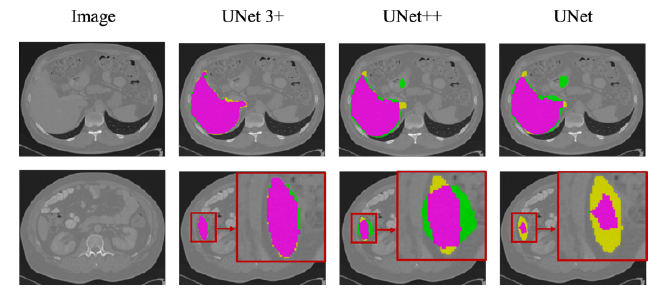
紫色区域:真阳性(TP);黄色区域:假阴性(FN);绿色区域:假阳性 (FP)。
UNet3+不仅能准确定位器官,还能产生连贯的边界,即使是在小物体的情况下。
3.3 与其他方法比较
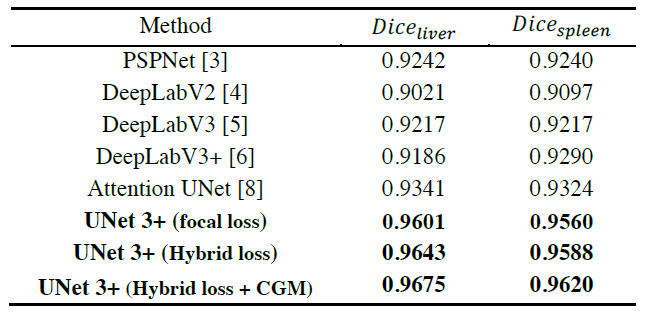
肝脏和脾脏数据集
所有结果直接来自单模型测试,不依赖任何后处理工具。
通过考虑像素级、补丁级、地图级优化,所提出的混合损失函数极大地提高了性能。
此外,UNet 3+利用分类指导模块(CGM),巧妙地避免了复杂背景下的过度分割。
最后,UNET 3+性能优于Attention UNET,PSPNet,DeepLabV2,DeepLabV3和DeepLabv3 +。
本文仅做学术分享,如有侵权,请联系删文。
下载1
在「3D视觉工坊」公众号后台回复:3D视觉,即可下载 3D视觉相关资料干货,涉及相机标定、三维重建、立体视觉、SLAM、深度学习、点云后处理、多视图几何等方向。
下载2
在「3D视觉工坊」公众号后台回复:3D视觉github资源汇总,即可下载包括结构光、标定源码、缺陷检测源码、深度估计与深度补全源码、点云处理相关源码、立体匹配源码、单目、双目3D检测、基于点云的3D检测、6D姿态估计源码汇总等。
下载3
在「3D视觉工坊」公众号后台回复:相机标定,即可下载独家相机标定学习课件与视频网址;后台回复:立体匹配,即可下载独家立体匹配学习课件与视频网址。
重磅!3DCVer-学术论文写作投稿 交流群已成立
扫码添加小助手微信,可申请加入3D视觉工坊-学术论文写作与投稿 微信交流群,旨在交流顶会、顶刊、SCI、EI等写作与投稿事宜。
同时也可申请加入我们的细分方向交流群,目前主要有3D视觉、CV&深度学习、SLAM、三维重建、点云后处理、自动驾驶、多传感器融合、CV入门、三维测量、VR/AR、3D人脸识别、医疗影像、缺陷检测、行人重识别、目标跟踪、视觉产品落地、视觉竞赛、车牌识别、硬件选型、学术交流、求职交流、ORB-SLAM系列源码交流、深度估计等微信群。
一定要备注:研究方向+学校/公司+昵称,例如:”3D视觉 + 上海交大 + 静静“。请按照格式备注,可快速被通过且邀请进群。原创投稿也请联系。

▲长按加微信群或投稿

▲长按关注公众号
3D视觉从入门到精通知识星球:针对3D视觉领域的视频课程(三维重建系列、三维点云系列、结构光系列、手眼标定、相机标定、orb-slam3等视频课程)、知识点汇总、入门进阶学习路线、最新paper分享、疑问解答五个方面进行深耕,更有各类大厂的算法工程人员进行技术指导。与此同时,星球将联合知名企业发布3D视觉相关算法开发岗位以及项目对接信息,打造成集技术与就业为一体的铁杆粉丝聚集区,近2000星球成员为创造更好的AI世界共同进步,知识星球入口:
学习3D视觉核心技术,扫描查看介绍,3天内无条件退款

圈里有高质量教程资料、可答疑解惑、助你高效解决问题
觉得有用,麻烦给个赞和在看~ 